1. はじめに (Introduction)
ChIP-seqとは?- エピジェネティクス解明の鍵 (What is ChIP-seq? – The Key to Unlocking Epigenetics)
私たちの体を作る設計図であるDNAは、全ての細胞でほぼ同じ配列を持っています。しかし、細胞の種類によって働く遺伝子が異なり、体の各部分が特有の機能を持つのはなぜでしょうか?この謎を解く鍵の一つが「エピジェネティクス」です。エピジェネティクスとは、DNAの塩基配列の変化を伴わずに遺伝子の働きを制御する仕組みのことであり、細胞の個性や発生、さらには病気の発症にも深く関わっています 1。
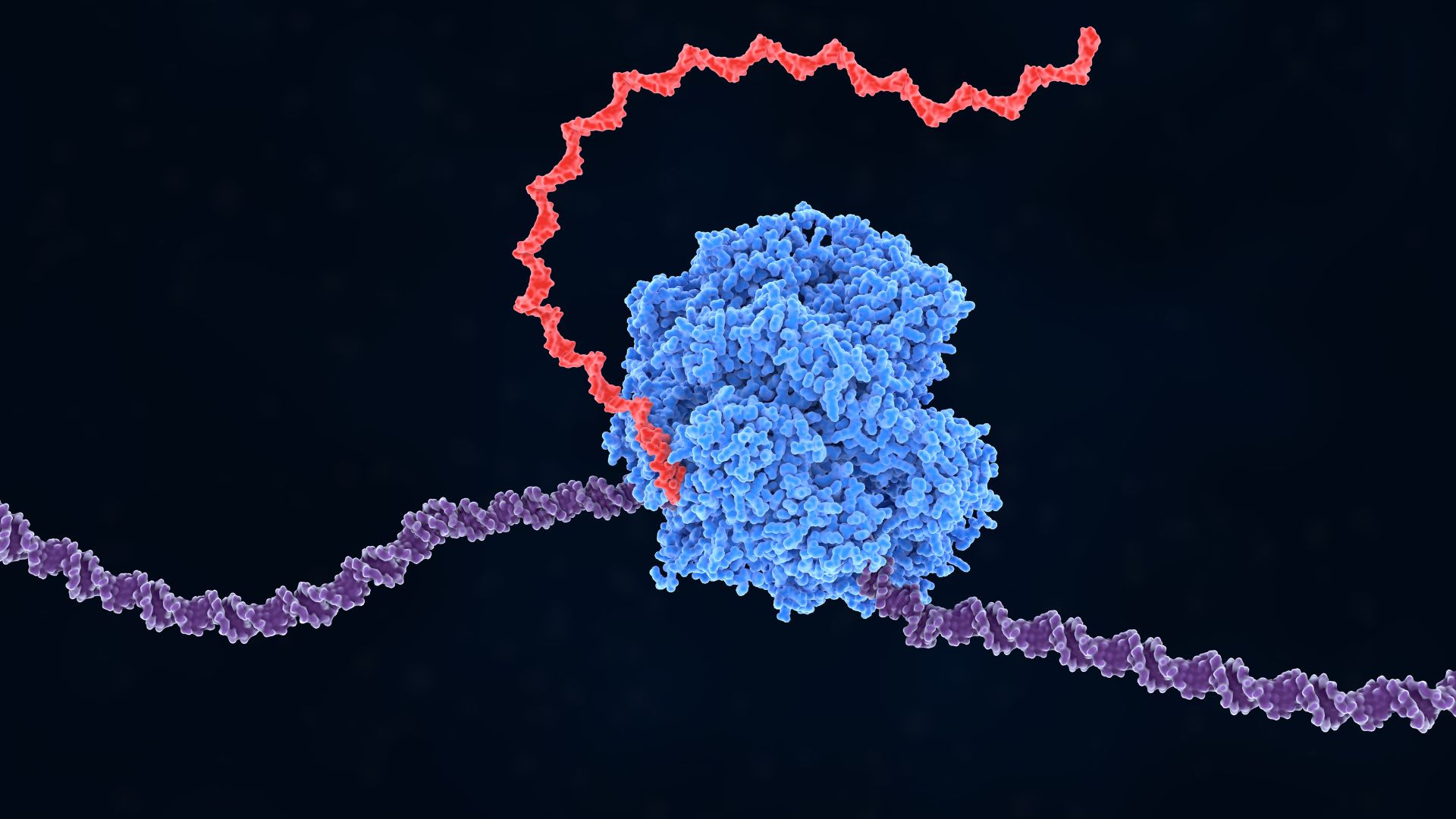
このエピジェネティクスのメカニズムをゲノムワイドに解明する強力な技術が、ChIP-seq (Chromatin Immunoprecipitation Sequencing、クロマチン免疫沈降シーケンシング) です 3。ChIP-seqは、特定のタンパク質がゲノムDNAのどの領域に結合しているか、あるいは特定のヒストン修飾がゲノム上のどこに存在するかを網羅的に明らかにすることができます 4。これにより、遺伝子発現のスイッチがどのようにオン・オフされるのか、細胞の運命がどのように決まるのかといった、生命の根源的な問いに迫る手がかりが得られます。
ChIP-seqは、クロマチン免疫沈降 (ChIP) という古典的な生化学的手法と、次世代シーケンサー (NGS) による網羅的なDNA配列解読技術を組み合わせたものです 3。2007年にRobertsonらによって哺乳類の生細胞内での転写因子のDNA結合部位を特定するために開発されて以来 1、ChIP-seq技術の進歩はクロマチン生物学と遺伝子制御の研究に革命をもたらしました 1。
この記事の目的と対象読者 (Purpose and Target Audience of This Article)
この記事は、ChIP-seq技術について学び始めたいと考えている初学者の方々を対象としています。生命科学系の学生、研究を始めたばかりの研究者、あるいは異分野からエピジェネティクス研究に興味を持った方々にとって、ChIP-seqの基本的な原理から最新の技術動向、そして今後の展望までを、国外の文献情報を中心に、わかりやすく解説することを目的としています。専門用語についても丁寧に説明し、複雑な実験の流れやデータ解析の概要を理解するための一助となることを目指します。
ChIP-seqを学ぶことの重要性 (Importance of Learning ChIP-seq)
ChIP-seqは、現代の生命科学研究において不可欠な基盤技術の一つとなっています。遺伝子の発現制御、細胞の分化や発生、がんをはじめとする様々な疾患のメカニズム解明など、幅広い研究分野で活用されています 1。ChIP-seqによって得られるゲノムワイドなタンパク質-DNA相互作用やヒストン修飾のマップは、生命現象をシステムレベルで理解するための貴重な情報源となります。
この技術を理解することは、エピジェネティクスというダイナミックな生命制御システムを深く探求するための第一歩です。また、ChIP-seqから派生した新しい技術も次々と開発されており、その進化は止まることを知りません。本記事を通じて、ChIP-seqの基礎を固め、エピジェネティクス研究の奥深さと面白さに触れていただければ幸いです。
2. ChIP-seqの基礎 – 原理と実験の流れを理解する (Fundamentals of ChIP-seq – Understanding the Principles and Experimental Workflow)
ChIP-seqを理解するためには、まずその基本原理と実験がどのようなステップで進められるのかを把握することが重要です。ここでは、ChIP-seqが何を明らかにする技術なのか、そして実験室での操作 (ウェット実験) とコンピュータ上での解析 (ドライ解析) がどのように行われるのかを解説します。
ChIP-seqの基本原理 – 何を明らかにする技術か (Basic Principle of ChIP-seq – What Does This Technology Reveal?)
ChIP-seqの基本的な目的は、特定のタンパク質がゲノムDNA上のどこに結合しているか、あるいは特定の化学修飾を受けたヒストンがゲノム上のどの領域に存在するかを、ゲノム全体にわたって明らかにすることです 3。
私たちの細胞核内では、DNAはヒストンと呼ばれるタンパク質に巻き付き、「クロマチン」という構造を形成しています 4。このクロマチン構造は、遺伝子のスイッチをオンにしたりオフにしたりする上で非常に重要な役割を果たします。転写因子のようなタンパク質が特定のDNA配列に結合することや、ヒストンがメチル化やアセチル化といった化学修飾を受けることは、遺伝子の読み取られやすさ(遺伝子発現)を変化させます 1。
ChIP-seqは、この「タンパク質とDNAの相互作用」または「特定のヒストン修飾が存在するゲノム領域」を、まるでゲノム上の地図のように描き出す技術です。具体的には、まず細胞内のタンパク質とDNAを化学的に固定(架橋)し、次に目的のタンパク質または修飾ヒストンに特異的に結合する抗体を用いて、それらが結合しているDNA断片ごと回収(免疫沈降)します。そして、回収されたDNA断片の塩基配列を次世代シーケンサーで大量に解読し、ゲノム上のどの位置に由来するのかを特定します 1。これにより、例えばある転写因子がどの遺伝子の制御領域に結合しているのか、あるいは活性型/抑制型のヒストン修飾がゲノムのどこに分布しているのかといった情報を、ゲノムワイドに得ることができます 4。
この情報は、遺伝子発現制御のメカニズム解明、エピジェネティックな制御パターンの同定、疾患関連遺伝子の機能解析など、多岐にわたる生命科学研究において極めて重要です 1。
ChIP-seq実験のステップ・バイ・ステップ解説 (Step-by-Step Explanation of the ChIP-seq Experiment)
ChIP-seqの実験は、大きく分けてウェット実験とドライ解析の2つのフェーズから構成されます 4。ここでは、まずウェット実験の主要なステップを順に見ていきましょう。
1. 架橋 (Crosslinking)
ChIPアッセイの最初のステップは、細胞内のタンパク質とDNAの複合体を安定化させるための「架橋」です 9。多くのタンパク質-DNA相互作用は一時的なものであり、細胞内ではタンパク質やDNAは絶えず動いています 9。そのため、特定の時点での相互作用を「スナップショット」として捉えるために、ホルムアルデヒドなどの化学物質を用いてタンパク質とDNAを共有結合で結びつけ、固定します 8。
ホルムアルデヒドはゼロレングスクロスリンカーであり、直接相互作用する2つの分子の架橋に適しています 9。より高次の相互作用や大きなタンパク質複合体を捉えるためには、EGSやDSGといったより長いクロスリンカーが併用されることもあります 9。架橋時間は非常に重要で、短すぎると効率的な架橋ができず、長すぎると後のクロマチン断片化や溶解が困難になることがあります 9。架橋反応は、適切な時間で停止させる(クエンチする)必要があります 9。一部のヒストン(例:H3、H4)とDNAの相互作用は非常に強固なため、架橋が不要な場合 (Native ChIPと呼ばれる) もあります 9。
2. 細胞溶解とクロマチン断片化 (Cell Lysis and Chromatin Fragmentation)
架橋後、細胞膜を界面活性剤ベースの溶解液で破壊し、細胞内容物を放出させ、架橋されたタンパク質-DNA複合体を可溶化します 9。タンパク質-DNA相互作用は主に核内で起こるため、細胞質タンパク質を除去することでバックグラウンドシグナルを低減し、感度を向上させることができます 9。架橋によって複合体は安定化されているため、界面活性剤や塩の存在下でも影響を受けにくいです 9。この段階では、タンパク質分解酵素阻害剤やリン酸化酵素阻害剤の使用が、複合体の完全性を保つために不可欠です 9。
次に、抽出されたゲノムDNAを、解析に適したより小さな断片(通常200~1000塩基対程度)に「断片化」します 8。DNAの断片化は、ChIPの解像度を得る上で非常に重要なステップです 8。断片化は主に以下の2つの方法で行われます。
- 超音波処理 (Sonication): 機械的な力でDNAをランダムにせん断します 10。ソニケーターという専用の機器が必要で、温度管理や処理時間の最適化が求められます 10。
- 酵素処理 (Enzymatic digestion): Micrococcal Nuclease (MNase) などの酵素を用いてDNAを切断します 10。この方法は再現性が高く、多検体処理に適していますが、酵素がヌクレオソーム間領域を優先的に切断する傾向があるため、完全にランダムな断片化とは言えません 8。
どちらの方法を用いる場合も、適切な断片長が得られるように条件を最適化することが重要です 9。
3. 免疫沈降 (Immunoprecipitation – IP)
断片化されたクロマチンの中から、目的のタンパク質(または特定の修飾を持つヒストン)とそれが結合したDNA断片だけを選択的に回収するステップが「免疫沈降」です 3。このステップでは、目的タンパク質に特異的に結合する「抗体」が鍵となります 1。
まず、クロマチン抽出液に目的タンパク質に対する抗体を加えます。抗体は目的タンパク質に結合し、抗体-タンパク質-DNA複合体を形成します。次に、プロテインAやプロテインGなどが結合した磁気ビーズやアガロースビーズを加えると、これらのビーズが抗体に結合し、複合体全体を物理的に捉えることができます 9。その後、ビーズを洗浄することで、非特異的に結合した他のタンパク質やDNA断片を除去し、目的の複合体を濃縮します 3。
抗体の特異性と品質は、ChIP-seqの結果の正確性を左右する最も重要な要素の一つです 1。質の低い抗体を使用すると、目的外のDNA断片が多く回収され、ノイズの高いデータとなってしまいます。
4. DNA精製とライブラリー調製 (DNA Purification and Library Preparation)
免疫沈降で回収された複合体から、タンパク質を除去し、DNAだけを精製します 8。まず、架橋を解くために加熱処理やプロテイナーゼKというタンパク質分解酵素による処理を行います(脱架橋)8。その後、フェノール・クロロホルム抽出や専用のスピンカラムなどを用いてDNAを精製します 9。
精製されたDNA断片は、次世代シーケンサーで配列解読を行うために「ライブラリー」と呼ばれる状態に加工されます 4。このライブラリー調製では、DNA断片の両端に「アダプター」と呼ばれる短い既知配列のDNAを結合させます。アダプターは、シーケンサーのフローセルへの結合や、PCRによる増幅、シーケンス反応の開始点として機能します 11。
5. 次世代シーケンシング (Next-Generation Sequencing – NGS)
調製されたDNAライブラリーは、次世代シーケンサー(NGS)を用いて塩基配列が決定されます 3。NGSは、数百万から数十億ものDNA断片の配列を同時に、並行して解読することができる革新的な技術です 3。ChIP-seqでは、免疫沈降によって濃縮されたDNA断片の配列を読むことで、目的のタンパク質がゲノム上のどの領域に結合していたのかを特定するための元データが得られます。得られる配列データは「リード」と呼ばれます 3。
ChIP-seqデータ解析の概要 – シーケンスデータから生物学的知見へ (Overview of ChIP-seq Data Analysis – From Sequence Data to Biological Insights)
NGSから得られた大量のリードデータは、そのままでは生物学的な意味を持ちません。これらの生データを解析し、タンパク質の結合部位を特定し、その生物学的な意義を考察するプロセスが「ドライ解析」または「バイオインフォマティクス解析」です 4。
1. クオリティコントロール (Quality Control – QC)
まず、シーケンシングで得られたリードデータの品質をチェックします 8。FastQCのようなツールを用いて、各リードの塩基ごとの品質スコア、アダプター配列の混入の有無、GC含量の偏りなどを評価します 8。低品質なリードやアダプター配列は、下流の解析に影響を与えるため、トリミング(除去)処理が行われます。
2. リードマッピング (Read Mapping)
品質チェックと前処理を経たリードを、参照ゲノム配列(リファレンスゲノム)上のどこに由来するのかを特定する作業が「マッピング」または「アライメント」です 8。BowtieやBWAといったマッピングツールが用いられ、各リードがゲノム上のどの染色体のどの位置に最もよく一致するかを決定します 8。マッピング結果はSAM/BAM形式のファイルとして出力されます 8。
マッピングの際には、ゲノム上に複数箇所存在する繰り返し配列に由来するリード(マルチマップリード)の扱いなどが考慮されます 14。一般的に、ユニークにマッピングされたリードの割合が高いほど、ライブラリーの質が良いとされます 15。
3. ピークコール (Peak Calling)
マッピングされたリードのゲノム上の分布を解析し、統計的に有意にリードが濃縮されている領域、すなわちタンパク質の結合部位候補を特定する作業が「ピークコール」です 3。MACS2などのピークコールツールが広く用いられています 8。
ピークコールでは、ChIPサンプル(抗体で免疫沈降したサンプル)のリード分布を、コントロールサンプル(Input DNAやIgGコントロールなど)のリード分布と比較します 4。コントロールサンプルは、免疫沈降操作やシーケンスにおけるバイアスを補正するために重要です。リードが局所的に集積し、コントロールと比較して有意に高い山(ピーク)を形成している領域が、タンパク質の結合部位として同定されます 4。ピークの形状は、結合タンパク質の種類によって異なり、転写因子のような場合はシャープなピーク、ヒストン修飾の場合はブロードなピークを示すことがあります 12。
4. ピークのアノテーションと下流解析 (Peak Annotation and Downstream Analysis)
同定されたピーク領域が、ゲノム上のどのような機能領域(プロモーター、エンハンサー、遺伝子間領域など)に位置するのか、また、どの遺伝子に関連しているのかを調べるのが「ピークアノテーション」です 4。
さらに、ピーク領域の情報を用いて様々な下流解析が行われます 4。
- モチーフ解析 (Motif Analysis): ピーク領域内のDNA配列を解析し、タンパク質が特異的に認識・結合する短いDNA配列パターン(結合モチーフ)を同定します 4。MEME-ChIPなどのツールが用いられます 13。
- ジーンオントロジー解析 (Gene Ontology Analysis): ピーク近傍の遺伝子群が、どのような生物学的プロセス、分子的機能、細胞構成要素に関連しているかを統計的に解析します 4。GREATなどのツールが用いられます 13。
- パスウェイ解析 (Pathway Analysis): ピークに関連する遺伝子が関与するシグナル伝達経路や代謝経路を同定します 16。
- 他のオミックスデータとの統合解析: RNA-seq(遺伝子発現解析)やATAC-seq(クロマチンアクセシビリティ解析)などの他のゲノムワイドデータと統合することで、タンパク質の結合が遺伝子発現やクロマチン状態に与える影響をより深く理解することができます 14。
これらの解析を通じて、ChIP-seqデータから生物学的に意義のある知見を引き出すことを目指します。
ChIP-seqで得られるデータの可視化 – ゲノムブラウザで見るDNA結合部位 (Visualizing ChIP-seq Data – Viewing DNA Binding Sites with a Genome Browser)
ChIP-seq解析で同定されたピーク(タンパク質の結合領域)は、ゲノムブラウザと呼ばれるツールを用いて視覚的に確認することができます 13。UCSC Genome BrowserやIntegrative Genomics Viewer (IGV) などが代表的なゲノムブラウザです 13。
ゲノムブラウザ上では、染色体上の特定領域におけるリードの分布がヒストグラムや密度プロットとして表示されます 16。ChIPサンプルでリードが集中して高い「山(ピーク)」を形成している領域が、タンパク質の結合部位を示唆します 17。コントロールサンプルと比較することで、そのピークが特異的な結合によるものか、あるいはバックグラウンドノイズなのかを判断するのに役立ちます 16。
ゲノムブラウザでの視覚化は、ピークコールの結果を検証したり、特定の遺伝子や領域における結合パターンを詳細に観察したりする上で非常に有効な手段です。また、複数のChIP-seqデータや他のゲノムデータ(遺伝子アノテーション、保存領域など)を重ねて表示することで、より多角的な解析が可能になります 19。
3. ChIP-seq技術の最前線 – 進化する手法と応用 (The Forefront of ChIP-seq Technology – Evolving Methods and Applications)
ChIP-seqはエピジェネティクス研究に革命をもたらしましたが、その技術も絶えず進化を続けています。従来型のChIP-seqが抱える課題を克服し、より高感度、高解像度、あるいは低細胞数での解析を可能にする新しい手法が次々と開発されています。
従来型ChIP-seqの課題と限界 (Challenges and Limitations of Conventional ChIP-seq)
従来型のChIP-seqは強力なツールである一方、いくつかの課題や限界も指摘されています 11。
- 抗体の特異性と品質: 結果の信頼性は使用する抗体の特異性に大きく依存します。非特異的な結合が多い抗体を使用すると、バックグラウンドノイズが高くなり、偽陽性のピークが検出される可能性があります 11。
- 架橋の効率とアーティファクト: ホルムアルデヒドによる架橋は、タンパク質-DNA複合体を安定化させる一方で、架橋効率のばらつきや、タンパク質同士の架橋によるアーティファクト(人工産物)を生じる可能性があります 11。過度な架橋は、可溶性タンパク質の意図しない回収を引き起こすこともあります 22。
- 必要細胞数の多さ: 一般的に数百万個単位の細胞が必要とされ、希少な細胞種や臨床検体からの解析が困難な場合があります 7。
- クロマチン断片化のばらつき: 超音波処理によるクロマチン断片化は、再現性や均一性の確保が難しく、結果に影響を与えることがあります 11。
- コストと時間、専門知識: 実験操作が煩雑で時間がかかり(3日以上かかることも)、シーケンシングコストも比較的高額です。また、データ解析には専門的なバイオインフォマティクスの知識が必要です 11。
- 低親和性・一過性の相互作用の検出: 結合力の弱いタンパク質や、短時間しか結合しないタンパク質の検出は難しい場合があります 11。
- 解像度の限界: 標準的なChIP-seqでは、結合部位の同定精度は数十から数百塩基対程度であり、塩基対レベルでの精密なマッピングは困難です 3。
これらの課題を克服するために、様々な改良法や新しいアプローチが開発されてきました。
進化するChIP-seq関連技術 – より高感度・高解像度へ (Evolving ChIP-seq Related Technologies – Towards Higher Sensitivity and Resolution)
近年、ChIP-seqの限界を克服し、より少ない細胞数で、より高い感度と解像度を実現する新しい技術が登場しています。ここでは代表的なものをいくつか紹介します。これらの技術は、ChIPの原理を応用しつつも、クロマチン断片化やライブラリー調製の方法に工夫を凝らしています。
CUT&RUN (Cleavage Under Targets and Release Using Nuclease)
CUT&RUNは、ChIP-seqの代替技術として注目されている手法で、特に低細胞数での解析や高解像度マッピングに優れています 26。この技術は、Steven Henikoffの研究室で開発されました 27。
- 原理: CUT&RUNでは、細胞を固定せず(ネイティブな状態)、透過処理した細胞や核をコンカナバリンAコーティング磁気ビーズに固定します 27。目的タンパク質に対する一次抗体、続いて二次抗体を反応させた後、プロテインAとMicrococcal Nuclease (MNase) の融合タンパク質 (pA-MNaseまたはpAG-MNase) を加えます 27。プロテインAが抗体に結合することで、MNaseが目的タンパク質の近傍にリクルートされます。その後、カルシウムイオンを加えてMNaseを活性化させると、目的タンパク質の結合部位周辺のDNAのみが選択的に切断されます 27。切断された短いDNA断片は細胞外へ放出され、これを回収してシーケンスライブラリーを調製します 26。
- 利点:
- 低細胞数: 数百~数千細胞からでも高品質なデータが得られ、ChIP-seqと比較して必要な細胞数が大幅に少ないです 26。
- 低バックグラウンド・高S/N比: 標的部位周辺のみDNAが切断・遊離されるため、非特異的なDNAの混入が少なく、バックグラウンドノイズが低減され、高いシグナル対ノイズ比 (S/N比) が得られます 26。
- 高解像度: MNaseによる精密な切断により、ChIP-seqよりも高い解像度で結合部位をマッピングできます 27。
- 短時間・低コスト: 架橋、クロマチン断片化、免疫沈降後の洗浄といった煩雑なステップが簡略化され、実験時間が短縮されます。また、必要なシーケンスリード数もChIP-seqより少ないため、コストも抑えられます 26。
- 欠点・課題:
- ネイティブクロマチン: 生細胞または核を用いるため、サンプルの保存や処理に注意が必要です。凍結融解に弱い場合があります 27。
- 抗体の適合性: ChIP-seqで実績のある抗体が必ずしもCUT&RUNに適しているとは限りません 27。
- MNaseのバイアス: MNaseは特定の配列を好みやすい性質があるため、そのバイアスを考慮する必要があります。
CUT&Tag (Cleavage Under Targets and Tagmentation)
CUT&Tagは、CUT&RUNをさらに発展させた技術で、同じくHenikoffの研究室で開発されました 31。特にライブラリー調製の簡便さと、さらなる低細胞数・シングルセル解析への適応性が特徴です。
- 原理: CUT&Tagもネイティブな細胞または核を用い、抗体で目的タンパク質を標識するまではCUT&RUNと類似しています 31。しかし、DNA切断とアダプター付加を同時に行う「タッグメンテーション」という反応を利用する点が異なります。具体的には、プロテインAとTn5トランスポゼースの融合タンパク質 (pA-Tn5) を使用します。このpA-Tn5は、あらかじめシーケンシング用のアダプターDNAを搭載しています 31。pA-Tn5が抗体を介して目的タンパク質の近傍にリクルートされた後、マグネシウムイオンを加えてTn5を活性化させると、標的DNAが切断されると同時にアダプター配列が挿入されます 31。これにより、ライブラリー調製が非常に簡便になります 31。
- 利点:
- 極低細胞数・シングルセル対応: CUT&RUNよりもさらに少ない細胞数(数十~数百細胞)から解析可能で、シングルセルCUT&Tag (scCUT&Tag) も実現されています 31。
- 迅速・簡便なライブラリー調製: タッグメンテーションにより、DNA断片化とアダプターライゲーションがワンステップで行われるため、実験時間が大幅に短縮され、操作も簡便です 31。
- 低コスト: 必要な細胞数、試薬、シーケンス量が少ないため、非常にコスト効率が良いです 31。
- 高S/N比: 特にヒストン修飾の解析において、高いS/N比が得られると報告されています 30。
- 欠点・課題:
- Tn5のバイアス: Tn5トランスポゼースはオープンクロマチン領域に挿入されやすいという性質があり、これがバイアスとなる可能性があります 30。このため、洗浄条件の最適化などが重要です 32。
- 転写因子の解像度: pA-Tn5複合体がpA-MNaseよりも大きいため、転写因子のような点状の結合部位の解像度はCUT&RUNに劣る場合があります 31。
- 抗体の品質依存性: 他の免疫沈降ベースの手法と同様に、抗体の品質が結果を大きく左右します 31。
ChIP-exo (ChIP with Exonuclease Treatment)
ChIP-exoは、ChIP-seqの解像度を大幅に向上させ、ほぼ塩基対レベルでタンパク質のDNA結合境界を特定することを目指した技術です 25。
- 原理: ChIPで免疫沈降したDNA-タンパク質複合体に対し、5’→3’方向に二本鎖DNAを分解するラムダエキソヌクレアーゼという酵素を作用させます 25。この酵素は、タンパク質が結合している箇所で消化が停止するため、タンパク質結合部位の境界ぎりぎりまでDNAが削られます 25。その後、残ったDNA断片の末端を決定しシーケンスすることで、結合部位を高精度に同定します 25。
- 利点:
- 超高解像度: タンパク質結合部位の境界をほぼ1塩基対の精度で決定できます 23。
- バックグラウンド低減: エキソヌクレアーゼ処理により、非特異的なDNA断片が除去されるため、バックグラウンドが低減されます 25。
- 低親和性結合の検出向上: バックグラウンドが低いため、効率の悪い結合イベントも検出しやすくなります 25。
- 欠点・課題:
- 実験操作の複雑さ: 複数の酵素処理ステップが必要で、実験プロトコルが複雑です 2。
- マルチクロスリンク部位の解釈: 一つの結合イベント内で複数の架橋点が存在する場合、複数の異なる結合イベントとして誤解釈される可能性があります 25。
- 抗体の必要性: 他のChIPベースの手法と同様、高品質な抗体が必要です 25。
低細胞数ChIP-seq (Low-Input ChIP-seq) とシングルセルChIP-seq (scChIP-seq)
従来のChIP-seqでは大量の細胞が必要でしたが、近年の技術開発により、より少ない細胞数、究極的には単一細胞レベルでの解析も可能になりつつあります。
- 低細胞数ChIP-seq: 数百~数万細胞といった少量のサンプルからChIP-seqを行うための様々なプロトコルが開発されています。これには、キャリアDNAの添加、ライブラリー調製法の改良(例: TAF-ChIP 35、ChIPmentation 35)、マイクロ流体デバイスの利用などが含まれます。EpigenTek社のEpiNext™ ChIP-Seq High-Sensitivity Kitなども、低細胞数からの解析をサポートする製品の一つです 11。これらの技術は、希少細胞集団や臨床検体など、サンプル量が限られる場合の解析に道を開きました 7。
TAF-ChIPは、免疫沈降中にTn5トランスポゼースによるタッグメンテーションを利用してクロマチンを断片化し、ライブラリー調製を簡便化することで、100細胞程度からの高品質なデータ取得を可能にしています 35。これは、従来のソニケーションによる断片化が困難な低細胞数サンプルにおいて特に有効です 35。 - シングルセルChIP-seq (scChIP-seq): 個々の細胞におけるタンパク質-DNA相互作用やヒストン修飾のプロファイルをゲノムワイドに解析する技術です 24。細胞集団の不均一性(ヘテロogeneity)を明らかにし、個々の細胞が持つエピジェネティックな個性を捉えることができます 1。scDrop-ChIPのようなマイクロ流体デバイスを用いた手法や、タッグメンテーションベースの手法 (例: scCUT&Tag) などが開発されています 24。
- 利点 (低細胞数・シングルセル共通):
- 希少細胞の解析: 幹細胞、特定のがん細胞、発生初期の細胞など、数が限られる細胞種の解析が可能です 35。
- 細胞不均一性の解明: 細胞集団内に存在する多様なエピジェネティック状態を持つサブポピュレーションを同定し、それぞれの特徴を明らかにできます 1。
- 欠点・課題 (特にシングルセル):
- データスパース性: 単一細胞から得られるDNA量は極めて少ないため、データがスパース(情報がまばら)になりがちです 20。
- 技術的ノイズ: 増幅バイアスやドロップアウト(本来存在するはずのシグナルが検出されない)といった技術的なノイズが大きくなる傾向があります 20。
- プロトコルの複雑さとコスト: 実験操作がより高度になり、特殊な装置や試薬が必要となる場合があり、コストも高くなることがあります 24。
- データ解析の難易度: スパースでノイズの多いデータを扱うため、より高度なバイオインフォマティクス解析技術が求められます 20。
これらの新しい技術は、それぞれに長所と短所があり、目的とするタンパク質の種類、利用可能な細胞数、求める解像度などに応じて使い分けられます。
【比較表】ChIP-seqとその関連技術 – 特徴・利点・欠点・応用 (Comparison Table: ChIP-seq and Related Technologies – Features, Advantages, Disadvantages, Applications)
以下に、本稿で紹介したChIP-seqおよびその主要な関連技術について、その特徴、主な利点、欠点・課題、主な応用、典型的な細胞数、解像度をまとめます。これにより、各技術の位置づけと使い分けについて理解を深めることができます。
| 技術名 (Technology) | 主な特徴 (Key Features) | 主な利点 (Key Advantages) | 主な欠点/課題 (Key Disadvantages/Challenges) | 主な応用 (Primary Applications) | 細胞数 (Typical Cell Input) | 解像度 (Typical Resolution) |
| ChIP-seq (従来型) | 架橋、ソニケーション/酵素断片化、抗体による免疫沈降、NGS | 確立された手法、多様なターゲットに対応、豊富な既存データ | 必要細胞数が多い、架橋アーティファクト、解像度限界 (~数100bp)、時間とコスト 11 | 標準的な転写因子 (TF)・ヒストン修飾マッピング 1 | 106~107 細胞 | 数十~数百 bp |
| CUT&RUN | ネイティブクロマチン、抗体誘導pA-MNaseによる標的切断・遊離 | 低細胞数、低バックグラウンド、高S/N比、高解像度、迅速 26 | ネイティブ素材の扱い、抗体の適合性、MNaseバイアス 27 | 低細胞数でのTF・ヒストン修飾マッピング、高解像度マッピング 26 | 102~105 細胞 | 数十 bp ~ 塩基対レベルに近づく |
| CUT&Tag | ネイティブクロマチン、抗体誘導pA-Tn5による標的タッグメンテーション (切断とアダプター挿入の同時進行) | 極低細胞数、シングルセル対応、迅速・簡便なライブラリー調製、低コスト、高S/N比 (特にヒストン修飾) 30 | Tn5バイアス (オープンクロマチン領域への偏り)、TF解析時の解像度がCUT&RUNに劣る可能性、抗体の品質依存性 30 | 極低細胞数・シングルセルでのヒストン修飾・TFマッピング 31 | 10~105 細胞 (バルク) | 数十 bp (ヒストン修飾) |
| ChIP-exo | ChIP後、ラムダエキソヌクレアーゼ処理によるDNA末端消化 | 超高解像度 (ほぼ塩基対レベル)、バックグラウンド低減 25 | 実験操作の複雑さ、マルチクロスリンク部位の解釈、抗体の必要性 2 | TF結合部位の精密マッピング、フットプリント解析 25 | 107~1010 細胞 (従来) | ほぼ塩基対レベル |
| 低細胞数ChIP-seq (例: TAF-ChIP) | 少細胞数に最適化されたChIPプロトコル (例: タッグメンテーション支援断片化) | 希少細胞からのデータ取得、従来法より少ない細胞数で解析可能 11 | 技術的ノウハウが必要、手法により特性が異なる、増幅バイアス 20 | 希少細胞集団におけるTF・ヒストン修飾マッピング 35 | 102~104 細胞 | ChIP-seqに準じる (手法による) |
| scChIP-seq (例: scCUT&Tag) | 単一細胞レベルでのChIP解析 | 細胞不均一性の解明、個々の細胞のエピゲノムプロファイリング 1 | データスパース性、技術的ノイズ、プロトコルの複雑さ・コスト、高度なデータ解析 20 | 細胞集団のヘテロogeneity解析、希少細胞の特性評価 32 | 1細胞~ | 手法による (CUT&Tagベースなど) |
この表は、各技術の概要を掴むためのものであり、実際の実験計画においては、詳細なプロトコルや最新の研究動向を確認することが不可欠です。特に新しい技術は急速に改良が進んでいるため、常に情報をアップデートしていく必要があります。
4. ChIP-seqの今後の展開 – エピジェネティクス研究の未来を拓く (The Horizon: Future Developments and Impact of ChIP-seq)
ChIP-seqとその関連技術は、エピジェネティクス研究の進展に大きく貢献してきましたが、その進化はまだ道半ばです。臨床応用への期待、技術的課題の克服、そしてAIなどの新しい解析技術との融合により、ChIP-seqは生命科学の未来をさらに切り拓いていくと期待されます。
臨床応用への期待 – 病気の診断・治療への道 (Expectations for Clinical Applications – The Path to Disease Diagnosis and Treatment)
ChIP-seqによって得られるエピジェネティックな情報は、疾患のメカニズム解明や新たな診断・治療法の開発に繋がる可能性を秘めています。
- 疾患メカニズムの解明: がんや生活習慣病、神経変性疾患など多くの疾患において、エピジェネティックな異常が関与していることが明らかになっています 1。ChIP-seqは、これらの疾患における特異的なヒストン修飾パターンや転写因子の異常な結合を同定し、疾患の発症や進行におけるエピジェネティクスの役割を解明する上で重要な役割を果たします 7。
- プレシジョン・メディシン(精密医療)の推進: 個々の患者さんのエピジェネティックな状態に基づいて最適な治療法を選択する「プレシジョン・メディシン」への貢献が期待されています 7。例えば、特定のエピジェネティックな変化を標的とする薬剤(エピジェネティックドラッグ)の効果を予測したり、治療応答性をモニタリングしたりするのにChIP-seqが利用できる可能性があります 7。
- 希少臨床検体への対応: 臨床現場で得られるサンプルは、生検組織や循環腫瘍細胞など、量が限られている場合が少なくありません 7。この課題に対応するため、低細胞数ChIP-seqやシングルセルChIP-seqといった、微量サンプルからでも解析可能な技術の開発が精力的に進められています 7。これらの技術の発展は、ChIP-seqの臨床応用を加速させる上で不可欠です。
- 診断・予後予測マーカーの探索: 特定の疾患に特徴的なエピジェネティックなパターン(エピジェネティックシグネチャー)を同定し、それを疾患の早期診断や予後予測のためのバイオマーカーとして利用する研究も進んでいます。
このように、ChIP-seq技術の臨床応用への道のりは、疾患のより深い理解と個別化医療の実現に向けた重要なステップと言えます。この流れは、技術開発の方向性にも大きな影響を与えており、より感度が高く、よりロバストで、そして実際の臨床現場で遭遇するような困難なサンプルにも適用可能な手法への需要が、技術革新を後押ししています。例えば、従来のChIP-seqでは「結合の有無」が主な情報でしたが、臨床応用を考えると「結合の強さや修飾の量」といった定量的情報がより重要になるため、定量的なChIP-seq法の開発も求められています 7。
技術的課題と克服への取り組み – さらなる進化を目指して (Technical Challenges and Efforts to Overcome Them – Aiming for Further Evolution)
ChIP-seq技術がさらに発展し、その応用範囲を広げていくためには、いくつかの技術的課題を克服する必要があります。
- 標準化と再現性の向上: ChIP-seq実験は多くのステップを含み、実験条件や解析パイプラインの選択によって結果が変動しうるため、プロトコルの標準化やデータ解析手法の統一化を進め、実験間の再現性や比較可能性を高めることが重要です 15。
- 抗体の品質向上: ChIP-seqの成功は、依然として使用する抗体の特異性と親和性に大きく依存します 11。より高品質で、多様な標的タンパク質や修飾に対応できる抗体の開発が継続的に求められています。
- 定量性の向上: 従来のChIP-seqは、タンパク質の結合やヒストン修飾の「有無」や「相対的な濃縮度」を評価するのが主でしたが、より正確にこれらの量を「絶対的」に定量する技術の開発が望まれています 7。これは、疾患状態における微妙なエピジェネティックな変化を捉えたり、薬剤の効果を精密に評価したりする上で重要となります。
- シングルセルChIP-seqの高度化: シングルセルレベルでのエピゲノム解析は、細胞の不均一性を理解する上で非常に強力ですが、データのスパース性、ノイズ、コストといった課題があります 20。これらの課題を克服し、よりロバストで簡便、かつ安価なシングルセルChIP-seq技術の開発が期待されます。
- 空間的情報の付加: 近年では、組織内での細胞の位置情報を保持したままエピゲノム情報を解析する「空間エピゲノミクス」という新しい分野も登場しています。ChIP-seq技術を空間解析と組み合わせることで、組織構築や疾患微小環境におけるエピジェネティック制御の理解が深まると期待されます。
これらの課題解決に向けた研究開発は、ChIP-seq技術をより洗練させ、新たな発見を促進することに繋がります。
AI・機械学習の活用 – 膨大なデータから新たな知見を掘り起こす (Utilizing AI and Machine Learning – Unearthing New Knowledge from Vast Data)
ChIP-seqやその他のオミックス技術によって生成されるデータは膨大であり、その複雑なパターンの中から生物学的に意味のある情報を見つけ出すためには、高度な計算科学的手法が不可欠です。特に、人工知能(AI)や機械学習(ML)の活用は、ChIP-seqデータ解析の新たな地平を切り拓くと期待されています。
- 大規模データ解析の効率化: AI/MLは、数百、数千といった多数のChIP-seqサンプルや、他のオミックスデータと統合された複雑なデータセットを効率的に処理し、解析する能力に長けています 14。
- 複雑なパターンの認識: ヒトの目では捉えきれないような、複数の転写因子やヒストン修飾の組み合わせによって形成される複雑なエピジェネティックな「状態」や制御ネットワークのパターンを、AI/MLはデータ駆動的に発見することができます 14。
- 予測モデリング: ChIP-seqで得られたエピジェネティック情報(転写因子の結合プロファイル、ヒストン修飾パターンなど)を入力として、遺伝子の発現レベルや疾患発症リスクなどを予測するモデルを構築することが可能です。
- 新規機能ゲノム領域のデノボアノテーション: 未知の機能を持つゲノム領域(例えば、新しいエンハンサーやサイレンサーなど)を、ChIP-seqデータなどのパターンからAI/MLを用いてデノボ(既知情報に頼らず)に予測し、アノテーションすることができます 12。
AI/ML技術の発展とChIP-seqデータの蓄積は、互いに補完し合いながら、これまで見過ごされてきた生命現象の制御原理の発見や、疾患メカニズムのより深い理解に貢献していくでしょう。
そして、ChIP-seqとその派生技術が臨床応用へと進み、AIによる解析が一般化するにつれて、エピゲノム情報という新たな個人情報に関する倫理的な側面も考慮する必要が生じてきます。エピゲノムは、遺伝情報(ゲノム)とは異なり、生活習慣や環境要因によって変化しうるダイナミックな情報です 1。これが疾患の診断や治療法の選択に用いられるようになると 7、その情報のプライバシー保護、解釈の妥当性、そして情報に基づく差別が生じないような社会的な枠組み作りが、技術の健全な発展と社会実装のために重要になってきます。これは、技術が成熟し、社会に広く浸透していく過程で必然的に生じる「波及効果」であり、初学者の段階から、技術の持つ可能性と同時に、その社会的な意味合いについても意識を向けることが望ましいでしょう。
初心者にとってのChIP-seq学習のポイント – 複雑な技術を理解するために (Learning Points for Beginners – Understanding Complex Technology)
ChIP-seqは多岐にわたる知識と技術を要するため、初学者にとっては複雑に感じられるかもしれません。しかし、以下のポイントを押さえることで、効率的に学習を進めることができます。
- 基本原理の理解: なぜ架橋するのか、なぜ免疫沈降するのか、シーケンシングで何がわかるのか、ピークとは何か、といった各ステップの「なぜ」を理解することが、技術全体を把握する上で最も重要です。
- コントロール実験の重要性の認識: ChIP-seqの結果は相対的なものであり、適切なコントロール実験(Input DNA、IgGコントロールなど)との比較によってはじめて意味を持ちます。コントロールの役割を理解しましょう。
- 限界と注意点の把握: 抗体の品質、必要な細胞数、解像度の限界など、ChIP-seqが抱える一般的な課題や注意点を認識しておくことで、実験計画や結果の解釈において適切な判断ができます。
- 基本的なデータ解析の流れを掴む: QC、マッピング、ピークコールといった基本的な解析ステップの目的と、そこで用いられる代表的なツール(FastQC, Bowtie/BWA, MACS2など)の名前と役割を覚えましょう。
- データの可視化に慣れる: ゲノムブラウザ (UCSC Genome Browser, IGVなど) を使って、実際のChIP-seqデータ(ピークの形状、コントロールとの比較など)を見てみることは、データのイメージを掴むのに非常に役立ちます。
- 公開データの活用: ENCODEプロジェクト 2 やGEO (Gene Expression Omnibus) といった公共データベースには、膨大なChIP-seqデータが登録されています。これらのデータをダウンロードして自分で解析してみたり、論文中の図と照らし合わせたりすることは、実践的な学習に繋がります。
- チュートリアルやトレーニングコースの利用: オンラインで利用可能なチュートリアル(例: Galaxy Project 29)や、学会・研究機関が提供するトレーニングコースに参加するのも良い方法です 17。
焦らず一つ一つのステップを理解し、実際にデータに触れる機会を増やすことが、複雑な技術を習得するための近道となるでしょう。
5. まとめ (Conclusion: ChIP-seq – A Key to Understanding Life’s Code)
ChIP-seqの重要性の再確認 (Reiteration of ChIP-seq’s Importance)
ChIP-seqは、特定のタンパク質とDNAの相互作用やヒストン修飾のゲノムワイドな分布を明らかにするための強力な技術であり、現代のゲノム科学およびエピジェネティクス研究において中心的な役割を担っています 1。この技術によって、遺伝子発現制御の複雑なネットワーク、細胞の運命決定のメカニズム、そして様々な疾患の分子的基盤に関する我々の理解は飛躍的に深まりました。
技術の進化と今後の展望 (Technological Evolution and Future Outlook)
ChIP-seqは、その登場以来、絶え間ない技術改良が重ねられてきました。CUT&RUN、CUT&Tag、ChIP-exoといった新しい手法は、より少ない細胞数で、より高い感度と解像度を実現し、これまで解析が困難であった生物学的問いへのアプローチを可能にしています 25。また、シングルセルレベルでのエピゲノム解析技術の発展は、細胞の不均一性という新たな次元の理解を深めつつあります 1。
今後、ChIP-seqとその関連技術は、臨床応用への展開がますます加速するとともに、AIや機械学習といった先進的なデータ解析技術との融合により、生命の設計図のさらなる深淵を探求し、基礎生物学の進展、革新的な創薬、そして個別化医療の実現に大きく貢献していくことが期待されます 7。
初学者へのメッセージ (Message for Beginners)
ChIP-seqは、一見すると複雑で難解に感じられるかもしれませんが、その基本原理を理解し、一つ一つのステップを丁寧に学んでいけば、必ずその奥深さと面白さを実感できるはずです。この記事が、皆さんがChIP-seqという魅力的な技術の世界へ足を踏み入れるための一助となれば幸いです。生命のコードを解き明かす旅は、まだ始まったばかりです。
6. 参考文献 (References)
- Robertson, G., Hirst, M., Bainbridge, M., Bilenky, M., Zhao, Y., Zeng, T.,… & Jones, S. J. (2007). Genome-wide profiles of STAT1 DNA association using chromatin immunoprecipitation and massively parallel sequencing. Nature methods, 4(8), 651-657. 1
- Park, P. J. (2009). ChIP–seq: advantages and challenges of a maturing technology. Nature reviews genetics, 10(10), 669-680.
- Nakato, R., & Shirahige, K. (2017). Recent advances in ChIP-seq analysis: from quality management to whole-genome annotation. Briefings in bioinformatics, 18(2), 279-290. 12
- Skene, P. J., & Henikoff, S. (2017). An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. eLife, 6, e21856. (CUT&RUN) 30
- Kaya-Okur, H. S., Wu, S. J., Codomo, C. A., Pledger, E. S., Bryson, T. D., Henikoff, J. G.,… & Henikoff, S. (2019). CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nature communications, 10(1), 1-10. (CUT&Tag) 30
- Rhee, H. S., & Pugh, B. F. (2011). Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell, 147(6), 1408-1419. (ChIP-exo) 25
- Kelsey, G., Stegle, O., & Reik, W. (2017). Single-cell epigenomics: Recording the dynamic epigenome. Genome Biology, 18(1), 1-15. (scChIP-seq関連のレビューとして)
- Zhang, Y., & Liu, T. (2014). Overview of ChIP-seq analysis. Current protocols in molecular biology, 105(1), 2-7. 15
(注: 上記は代表的な文献であり、本文中の引用箇所と合わせてご参照ください。実際の論文リストは、より広範な文献調査に基づいて作成する必要があります。)
7. 関連キーワード (Related Keywords for SEO)
ChIP-seq, クロマチン免疫沈降, 次世代シーケンサー, NGS, エピジェネティクス, ヒストン修飾, 転写因子, DNA結合タンパク質, 遺伝子発現調節, ピークコール, モチーフ解析, CUT&RUN, CUT&Tag, scChIP-seq, ChIP-exo, ゲノム解析, 初心者向け, 原理, 方法, 実験手順, データ解析, 最新技術, 臨床応用, エピゲノム, ChIP-seq やり方, ChIP-seq 解析ソフト, ChIP-seq コスト, ChIP-seq データ 解釈. 2
引用文献
- ChIP-Sequencing: Deciphering Epigenetic Modifications and Gene Regulation, 5月 18, 2025にアクセス、 https://www.azolifesciences.com/article/ChIP-sequencing-Deciphering-Epigenetic-Modifications-and-Gene-Regulation.aspx
- tohoku.repo.nii.ac.jp, 5月 18, 2025にアクセス、 https://tohoku.repo.nii.ac.jp/record/132854/files/200325-Anzawa-719-1.pdf
- ChIP sequencing – Wikipedia, 5月 18, 2025にアクセス、 https://en.wikipedia.org/wiki/ChIP_sequencing
- A beginner’s guide to ChIP-seq analysis | The Biochemist | Portland …, 5月 18, 2025にアクセス、 https://portlandpress.com/biochemist/article/47/1/21/235716/A-beginner-s-guide-to-ChIP-seq-analysis
- 少数細胞エピゲノム解析技術の開発 – 日本生化学会, 5月 18, 2025にアクセス、 https://seikagaku.jbsoc.or.jp/10.14952/SEIKAGAKU.2021.930872/data/index.html
- ChIP-seq法 – 羊土社, 5月 18, 2025にアクセス、 https://www.yodosha.co.jp/jikkenigaku/keyword/index.html?id=696
- ChIP-seq in studying epigenetic mechanisms of disease and …, 5月 18, 2025にアクセス、 https://pubmed.ncbi.nlm.nih.gov/27319740/
- ChIP-Seq: Introduction, Features, Workflow, and Applications – CD …, 5月 18, 2025にアクセス、 https://rna.cd-genomics.com/resource/chip-seq-introduction-features-workflow-and-applications.html
- A Step-by-Step Guide to Successful Chromatin Immunoprecipitation …, 5月 18, 2025にアクセス、 https://www.thermofisher.com/us/en/home/life-science/antibodies/antibodies-learning-center/antibodies-resource-library/antibody-application-notes/step-by-step-guide-successful-chip-assays.html
- Chromatin IP (ChIP Assays) | Thermo Fisher Scientific – US, 5月 18, 2025にアクセス、 https://www.thermofisher.com/us/en/home/life-science/protein-biology/protein-biology-learning-center/protein-biology-resource-library/pierce-protein-methods/chromatin-ip-chip-assays.html
- ChIP-Seq – Uses & Challenges | EpigenTek, 5月 18, 2025にアクセス、 https://www.epigentek.com/catalog/chip-seq-uses-challenges-n-145.html?newsPath=16
- Recent advances in ChIP-seq analysis: from quality management to whole-genome annotation – ENS, 5月 18, 2025にアクセス、 https://www.bio.ens.psl.eu/~mthomas/other/chip-seq-training/articles/2016_Nakato_Brief_Bioinformatics.pdf
- A step-by-step guide to ChIP-seq data analysis – Abcam, 5月 18, 2025にアクセス、 https://www.abcam.com/en-us/webinars/a-step-by-step-guide-to-chip-seq-data-analysis
- Recent advances in ChIP-seq analysis: from quality management to …, 5月 18, 2025にアクセス、 https://academic.oup.com/bib/article/18/2/279/2453282
- Computational methodology for ChIP-seq analysis – PMC, 5月 18, 2025にアクセス、 https://pmc.ncbi.nlm.nih.gov/articles/PMC4346130/
- ChIP-Seq Analysis Tutorial – Basepair, 5月 18, 2025にアクセス、 https://www.basepairtech.com/knowledge-center/chip-seq-analysis-tutorial/
- biosciencedbc.jp, 5月 18, 2025にアクセス、 https://biosciencedbc.jp/gadget/human/20150806_morioka.pdf
- ChIP-seq analysis – ENS, 5月 18, 2025にアクセス、 https://www.bio.ens.psl.eu/~mthomas/other/chip-seq-training/
- How to Integrate ChIP-Seq and RNA-Seq Data – Partek – Illumina, 5月 18, 2025にアクセス、 https://help.partek.illumina.com/partek-flow/blog-posts/how-to-integrate-chip-seq-and-rna-seq-data
- Integrating ChIP-seq with other functional genomics data | Briefings …, 5月 18, 2025にアクセス、 https://academic.oup.com/bfg/article/17/2/104/4944665
- ChIP-Seq – CD Genomics, 5月 18, 2025にアクセス、 https://www.cd-genomics.com/chip-seq.html
- A beginner’s guide to ChIP-seq analysis | The Biochemist | Portland …, 5月 18, 2025にアクセス、 https://portlandpress.com/biochemist/article/doi/10.1042/bio_2024_920/235716/A-beginner-s-guide-to-ChIP-seq-analysis
- ChIP-seq Advantages and Limitations, 5月 18, 2025にアクセス、 https://www.news-medical.net/life-sciences/ChIP-seq-Advantages-and-Limitations.aspx
- ChIP Sequencing (ChIP-seq): Principle, Steps, Uses, Diagram, 5月 18, 2025にアクセス、 https://microbenotes.com/chip-sequencing/
- ChIP-exo – Wikipedia, 5月 18, 2025にアクセス、 https://en.wikipedia.org/wiki/ChIP-exo
- CUT&RUN Overview – EpiCypher, 5月 18, 2025にアクセス、 https://www.epicypher.com/resources/cut-and-run-overview/
- www.activemotif.com, 5月 18, 2025にアクセス、 https://www.activemotif.com/documents/CUT-RUN-Complete-Guide.pdf
- DNA binding sites map : CUT & RUN vs CUT & Tag – France …, 5月 18, 2025にアクセス、 https://www.france-genomique.org/technological-expertises/regulome/cartographie-des-contacts-de-la-chromatine-cutrun/?lang=en
- Epigenetics / CUT&RUN data analysis / Hands-on – Galaxy Training …, 5月 18, 2025にアクセス、 https://training.galaxyproject.org/training-material/topics/epigenetics/tutorials/cut_and_run/tutorial.html
- Benchmark of chromatin–protein interaction methods in … – Frontiers, 5月 18, 2025にアクセス、 https://www.frontiersin.org/articles/10.3389/fcell.2025.1572405/full
- Efficient low-cost chromatin profiling with CUT&Tag – PMC, 5月 18, 2025にアクセス、 https://pmc.ncbi.nlm.nih.gov/articles/PMC8318778/
- (PDF) The development and application of cleavage under targets …, 5月 18, 2025にアクセス、 https://www.researchgate.net/publication/383843221_The_development_and_application_of_cleavage_under_targets_and_tagmentation_CUTTag_technology
- Comprehensive Guide to Understanding and Using CUT&Tag Assays, 5月 18, 2025にアクセス、 https://www.activemotif.com/blog-cut-tag
- Cut&tag: a powerful epigenetic tool for chromatin profiling – PMC, 5月 18, 2025にアクセス、 https://pmc.ncbi.nlm.nih.gov/articles/PMC10730171/
- TAF-ChIP: an ultra-low input approach for genome-wide chromatin …, 5月 18, 2025にアクセス、 https://pmc.ncbi.nlm.nih.gov/articles/PMC6653780/
- (PDF) Single Cell Sequencing, Its Application and Future Challenges, 5月 18, 2025にアクセス、 https://www.researchgate.net/publication/359395553_Single_Cell_Sequencing_Its_Application_and_Future_Challenges
- Single-Cell RNA Sequencing Frequently Asked Questions | GENEWIZ, 5月 18, 2025にアクセス、 https://web.genewiz.com/faqs/single-cell-rna-seq